Trotz deutlicher Verbesserung der Gewässergüte vieler bayerischer Fließgewässer besteht weiterhin eine zum Teil erhebliche Belastung mit Fäkalbakterien, die eine Nutzung als Badegewässer aus hygienischer Sicht einschränkt. Fäkalbakterien werden (a) durch die Einleitung von Abwässern zum Beispiel aus Kläranlagen oder Regenentlastungsbauwerken und (b) aus diffusen Quellen zum Beispiel durch landwirtschaftliche Aktivitäten oder Wildtiere in die Gewässer eingetragen. Die Kenntnis der Herkunft fäkaler Verunreinigungen ist deshalb entscheidend für Effizienz und Auswahl von Abhilfemaßnahmen für eine Verbesserung der hygienischen Situation im Gewässer. So sind teure Maßnahmen zur Abwasserdesinfektion nur dann sinnvoll, wenn der Eintrag von Bakterien – und damit auch möglicher Krankheitserreger – zum überwiegenden Teil tatsächlich von Kläranlageneinleitungen herrührt.
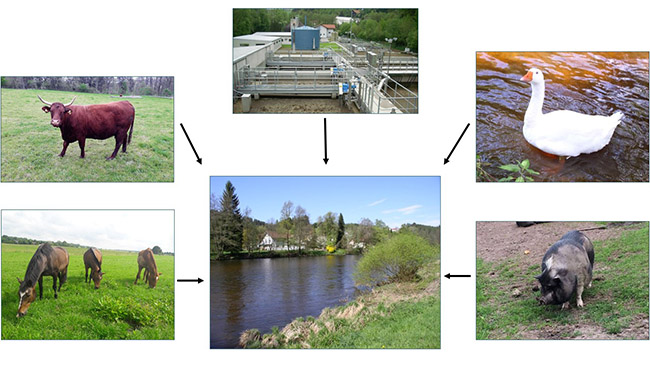 Mögliche Eintragsquellen fäkaler Verunreinigungen in ein Gewässer
Mögliche Eintragsquellen fäkaler Verunreinigungen in ein GewässerAnalytik
Der Nachweis von Fäkalindikatorbakterien (E. coli, Intestinale Enterokokken) gibt zwar einen Hinweis auf eine fäkale Belastung des Gewässers, erlaubt jedoch keinen Rückschluss auf deren Ursprung. Mit Hilfe sogenannter "Source tracking"-Methoden ist es mittlerweile möglich, der Herkunft solcher Verunreinigungen auf die Spur zu kommen. Eine molekularbiologische Methode wird am LfU als innovatives Verfahren in der Umweltanalytik eingesetzt. Dabei werden Umweltproben auf definierte DNA-Abschnitte von Darmbakterien untersucht, die speziell nur in menschlichem oder tierischem Kot vorkommen. Neben dem qualitativen Nachweis können diese sog. "DNA-Marker" mittels Real-Time-PCR-Analytik und Droplet Digital PCR auch quantifiziert werden.
 Droplet-Digital PCR: Analysengeräte (links) und Verteilung positiver und negativer Droplets (rechts)
Droplet-Digital PCR: Analysengeräte (links) und Verteilung positiver und negativer Droplets (rechts)Aktueller Kenntnisstand
Die bisherigen Untersuchungen zeigen, dass Microbial Source Tracking mit wirtsspezifischen DNA-Markern für den Einsatz in der Praxis geeignet ist. Zusammen mit der Betrachtung der örtlichen Gegebenheiten ist Microbial Source Tracking ein hilfreiches Werkzeug bei der Ursachenforschung und der Ermittlung der Herkunft fäkaler Gewässerbelastungen. Damit trägt es wesentlich zu einer zielorientierten Bewirtschaftung des Gewässers bei.
An der Ilz konnte mit Hilfe dieser Methode beispielsweise gezeigt werden, dass die mikrobiologisch-hygienische Belastung unter trockenen Wetterbedingungen fast ausschließlich auf vom Menschen stammende Fäkaleinträge – zum Beispiel aus Kläranlageneinleitungen - zurückzuführen ist. Starke Niederschläge führten zu einer Verschlechterung der mikrobiologisch-hygienischen Wasserqualität, die häufig mit einem Anstieg der Konzentration an humanbürtigen DNA-Markern verbunden war. Hier spielen sehr wahrscheinlich Einleitungen aus Regenentlastungsbauwerken eine Rolle, die bei hohen Regenintensitäten einen Teil des Abwassers in das Gewässer entlasten. Gleichzeitig wurden aber auch erhebliche Mengen eines DNA-Markers gemessen, der spezifisch für Wiederkäuer-Fäkalien ist. Bei Regenwetter sind in diesem Einzugsgebiet neben Einträgen aus dem Siedlungsbereich auch diffuse Belastungen aus landwirtschaftlich genutzten Flächen für die fäkale Verschmutzung von Bedeutung.