Genetische Verfahren im biologischen Monitoring
Die Artenvielfalt und Artzusammensetzung spielt für die Bewertung der Qualität von Fließgewässern eine zentrale Rolle. Im Bewertungsverfahren nach Wasserrahmenrichtlinie (WRRL) wird die Bestimmungsarbeit klassisch durch die Erkennung artspezifischer morphologischer Merkmale vorgenommen. Morphologische Bestimmungsarbeiten erfordern eine intensive Einarbeitung und viel Erfahrung und können bei schwer bestimmbaren Arten sehr zeitaufwendig sein. Nicht immer ist für alle Entwicklungsstadien eine eindeutige Artbestimmung anhand morphologischer Merkmale möglich. Genetische Verfahren ermöglichen es, Taxa anhand der DNA-Sequenz eines bestimmten Markergens molekularbiologisch zu bestimmen. Die Anwendung von genetischen Verfahren zur Identifikation von Arten hat durch die Entwicklung von Hochdurchsatz-Sequenzierung (Next Generation Sequencing) , die eine schnelle Analyse von Umweltproben ermöglicht, in den letzten Jahren in verschiedensten Bereichen enorm an Bedeutung gewonnen. Auch für die Bewertungsverfahren für Oberflächengewässer nach WRRL könnten die Fortschritte der genetischen Verfahren eine große Bereicherung sein, um Proben schnell und standardisiert zu analysieren. Vor diesem Hintergrund stellt sich die Frage, ob die aktuellen genetischen Verfahren bereits heute das Potential haben, das klassische Bewertungsverfahren gemäß Wasserrahmenrichtlinie in Hinblick auf die Makrozoobenthos (MZB) Bewertung für Fließgewässer zu optimieren bzw. zu ergänzen oder diese langfristig eventuell sogar zu ersetzen.
Wie wurden die Verfahren verglichen?
Um die Verfahren zu vergleichen, wurden im Rahmen dieser Studie für das LfU-Projekt "Monitoring Biologie – Anpassung an neue Herausforderungen" insgesamt 60 von der bayerischen Wasserwirtschaft erhobene und klassisch (morphologisch) bestimmte Makrozoobenthos Proben aus verschiedenen Fließgewässertypen mittels DNA Metabarcoding analysiert. Der Vorteil von Metabarcoding besteht darin, dass einzelne Arten aus einer Organismus Sammelprobe identifiziert werden können. Dazu wird die DNA aus der Sammelprobe extrahiert, das spezifische taxonomische Markergen für Makrozoobenthos mittels Polymerase-Kettenreaktion (PCR) vervielfältigt und durch Hochdurchsatz-Sequenzierung sequenziert. Diese Gensequenzen werden anschließend entsprechend ihrer DNA Sequenz Ähnlichkeit zu sogenannten OTUs (Operational taxonomic units) gruppiert. Diese OTU-Cluster werden schließlich mit Gensequenzen in Referenzdatenbanken (z.B. Barcode Of Life Database (BOLD)) verglichen und taxonomisch zugeordnet (Bioinformatische Auswertung). Die morphologisch bestimmten und die genetisch erstelltenTaxalisten wurden anschließend taxonomisch angeglichen und die Befunde verglichen. Die jeweils aus den Befundlisten resultierenden Bewertungsergebnisse (1: sehr gut, 2: gut, 3: mäßig, 4: unbefriedigend oder 5: schlecht) der Module "Saprobie" und "Allgemeine Degradation" , sowie der Gesamtbewertung der biologischen Qualitätskomponente Makrozoobenthos wurden ebenfalls verglichen. Eine Übersicht des methodischen Vorgehens ist in Abbildung 1 dargestellt.
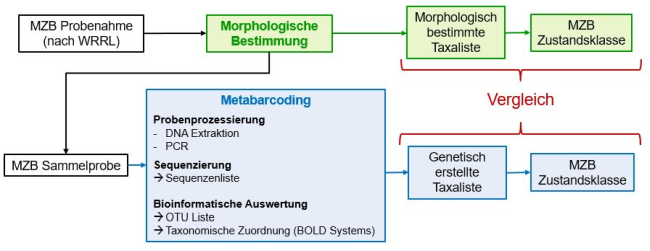 Abbildung 1: Übersicht des methodischen Vorgehens. Makrozoobenthos (MZB) Proben aus dem biologischen Monitoring wurden morphologisch bestimmt und mittels Metabarcoding genetisch analysiert. Das Metabarcoding umfasst dabei die Extraktion der DNA des spezifischen Markergens und deren Vervielfältigung mittels Polymerase-Kettenreaktion (polymerase chain reaction (PCR)) (Probenprozessierung). Die anschließende Sequenzierung und die abschließende taxonomische Zuordnung der gruppierten OTUs (operational taxonomic units) erfolgt über einen Abgleich der Sequenzen mit den in Referenzdatenbanken hinterlegten Sequenzen (bioinformatische Auswertung). Die daraus resultierenden Taxalisten und Bewertungsergebnisse (MZB Zustandsklasse) wurden anschließend miteinander verglichen.
Abbildung 1: Übersicht des methodischen Vorgehens. Makrozoobenthos (MZB) Proben aus dem biologischen Monitoring wurden morphologisch bestimmt und mittels Metabarcoding genetisch analysiert. Das Metabarcoding umfasst dabei die Extraktion der DNA des spezifischen Markergens und deren Vervielfältigung mittels Polymerase-Kettenreaktion (polymerase chain reaction (PCR)) (Probenprozessierung). Die anschließende Sequenzierung und die abschließende taxonomische Zuordnung der gruppierten OTUs (operational taxonomic units) erfolgt über einen Abgleich der Sequenzen mit den in Referenzdatenbanken hinterlegten Sequenzen (bioinformatische Auswertung). Die daraus resultierenden Taxalisten und Bewertungsergebnisse (MZB Zustandsklasse) wurden anschließend miteinander verglichen.Ist das Bestimmungsniveau vergleichbar?
Bei der Kontrolle der Ausbeute und Qualität der DNA hat sich gezeigt, dass die DNA einiger Proben stark degradiert war. Dies verdeutlicht die Notwendigkeit einer standardisierten und hinsichtlich DNA Konservierung optimierten Anleitung für die Probenahme und Handhabung. Nichtsdestotrotz zeigt der direkte Vergleich der morphologisch bestimmten und genetisch erstelltenTaxalisten, dass die Übereinstimmung besonders auf Gattungs- und Familienniveau hoch ist (Art: 61%, Gattung: 69%, Familie 78%, Ordnung 66%) (Abbildung 2). Von den morphologisch bestimmten Arten war die Wiederfindungsquote in den genetischen Befunden bei den Gliederfüßern (Arthropoda) am höchsten (70 %), gefolgt von den Ringelwürmern (Annelida) (41%). Bei den Weichtieren (Mollusca) wurde die geringste Übereinstimmung (24 %) zwischen beiden Verfahren erzielt, wohingegen Plattwürmer (Plathelminthes) genetisch nicht nachgewiesen wurden (Abbildung 3). Von den Gliederfüßern wurden deutlich mehr Taxa genetisch identifiziert als morphologisch bestimmt. Die Übereinstimmung der morphologisch bestimmten Arten mit den genetisch detektierten Taxa ist bei der Insecta Ordnung Trichoptera mit 77% am höchsten, gefolgt von Plecoptera (73%), Coleoptera (67%) und Diptera (66%). Die geringste Übereinstimmung liegt bei den Ephemeroptera vor (60%) (Abb. 4). Von den Insecta Ordnungen Plecoptera (Steinfliegen), Trichoptera (Köcherfliegen), Diptera (Zweiflügler) und Coleoptera (Käfer) wurden insgesamt mehr Arten durch das genetische Verfahren nachgewiesen (Abb. 4). Die genetische Artenvielfalt ist dabei bei Plecoptera, aber vor allem bei den Diptera im Vergleich zur morphologischen Bestimmung deutlich am größten. Der größte Teil der ausschließlich genetisch detektierten Diptera Taxa gehört zu der Familie der Chironomidae. Dies liegt daran, dass einige Arten dieser Ordnungen als Larve morphologisch nicht sicher differenziert werden können und daher auf niedrigere taxonomische Level bestimmt werden. Einige morphologisch bestimmte Taxa der Trichoptera (insbesondere der Familien Leptoceridae und Limnephilidae) und der Ephemeroptera (insbesondere der Familie Heptageniidae) wurden genetisch nicht nachgewiesen. Die Qualität der Artidentifizierung und Wiederfindung im genetischen Verfahren ist u.a. von dem gewählten Markergen sowie Primer Effizienz, aber auch von den hinterlegten Referenzen der verfügbaren Datenbanken abhängig und wird von Informationslücken und Fehlerquellen beeinträchtigt. So konnten zum Zeitpunkt dieser Studie wenige Arten nicht eindeutig zugeordnet werden, da der Artname in der Referenzdatenbank nicht hinterlegt war. Abgesehen von möglichen Fehlerquellen bei der morphologischen Bestimmung, besteht bei genetisch sehr ähnlichen Arten zudem eine hohe Verwechslungsgefahr bei der automatisierten taxonomischen Zuordnung.
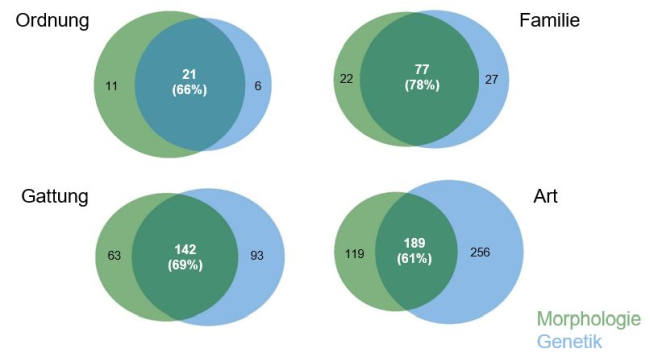 Abbildung 2: Übersicht der ausschließlich morphologisch (grün) und genetisch (blau) sowie gemeinsam identifizierten taxonomischen Level auf Ordnungs-, Familien-, Gattungs- und Artniveau
Abbildung 2: Übersicht der ausschließlich morphologisch (grün) und genetisch (blau) sowie gemeinsam identifizierten taxonomischen Level auf Ordnungs-, Familien-, Gattungs- und Artniveau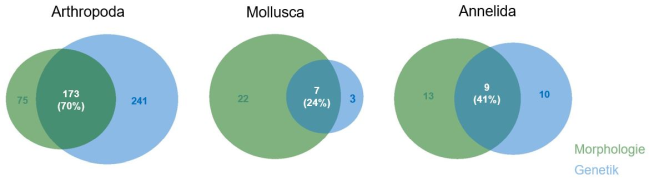 Abbildung 3: Anteile der ausschließlich morphologisch (grün) und genetisch (blau) sowie gemeinsam identifizierten Taxa der Stämme Arthropoda, Mollusca und Annelida
Abbildung 3: Anteile der ausschließlich morphologisch (grün) und genetisch (blau) sowie gemeinsam identifizierten Taxa der Stämme Arthropoda, Mollusca und Annelida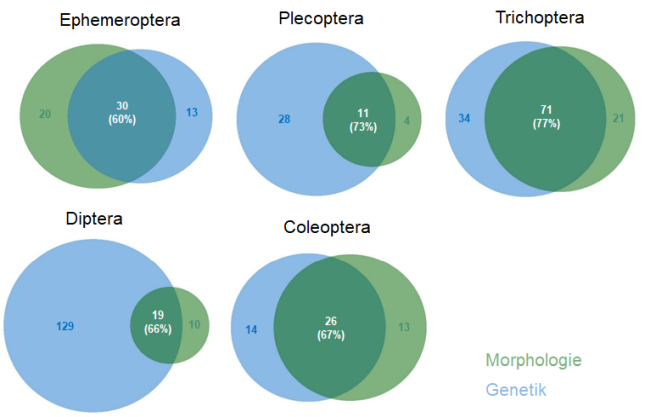 Abbildung 4: Anteile der ausschließlich morphologisch (grün) und genetisch (blau) sowie gemeinsam identifizierten Taxa der Insecta Ordnungen Ephemeroptera, Plecoptera, Trichoptera, Diptera und Coleoptera
Abbildung 4: Anteile der ausschließlich morphologisch (grün) und genetisch (blau) sowie gemeinsam identifizierten Taxa der Insecta Ordnungen Ephemeroptera, Plecoptera, Trichoptera, Diptera und ColeopteraErgeben sich Unterschiede in den Bewertungsergebnissen?
Durch genetische Verfahren können bislang keine Abundanzwerte (Anzahl der Individuen einer Art) der Taxa in der Probe ermittelt werden, so dass lediglich angegeben werden kann, ob ein bestimmtes Taxon in der Probe vorhanden ist (anstatt der Abundanz wird lediglich eine 1 für "anwesend" oder 0 für "nicht anwesend" angegeben). Folglich können Diskrepanzen in den Bewertungsergebnissen nicht nur durch Unterschiede zwischen den morphologischen und genetischen Befunden entstehen, sondern auch durch Berechnungen basierend auf Anwesenheitsdaten anstatt Abundanzwerte. Es wurden deshalb auch Berechnungen mit der morphologisch bestimmten Taxaliste durchgeführt, wobei die Abundanzwerte in dieser Liste durch eine 1 für "anwesend" oder eine 0 für "abwesend" ersetzt wurden (M01). Die Bewertungsergebnisse der klassischen (MA) und genetischen Verfahren (G) stimmen insgesamt gut überein, was die statistisch hoch signifikanten Korrelationen verdeutlichen (Abbildung 5).
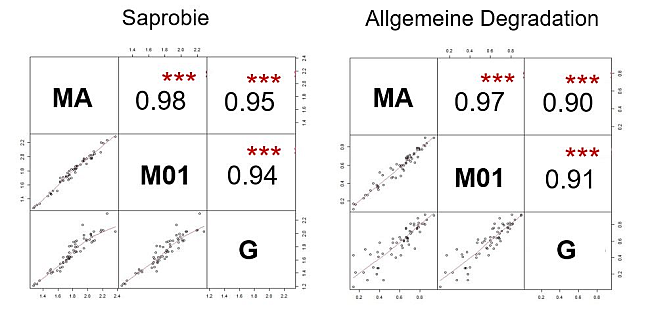 Abbildung 5: Streudiagramme (unterhalb der Diagonale) und Spearman-Korrelationskoeffizienten (oberhalb der Diagonale) der berechneten Bewertungsergebnisse der Makrozoobenthos Module "Saprobie" und "Allgemeine Degradation" basierend auf morphologisch und genetisch ermittelten Makrozoobenthos Befunden. Auf der Diagonalen sind die unterschiedlichen Berechnungsverfahren dargestellt (MA: Ergebnisse basieren auf Berechnung mit morphologisch bestimmten Taxalisten mit Abundanzwerten, M01: Ergebnisse basieren auf Berechnung mit morphologisch bestimmten Taxalisten mit An-/Abwesenheitsdaten, G: Ergebnisse basieren auf Berechnung mit genetisch erstellten Taxalisten mit An-/Abwesenheitsdaten). Statistisch hoch signifikante Korrelationen sind durch *** gekennzeichnet.
Abbildung 5: Streudiagramme (unterhalb der Diagonale) und Spearman-Korrelationskoeffizienten (oberhalb der Diagonale) der berechneten Bewertungsergebnisse der Makrozoobenthos Module "Saprobie" und "Allgemeine Degradation" basierend auf morphologisch und genetisch ermittelten Makrozoobenthos Befunden. Auf der Diagonalen sind die unterschiedlichen Berechnungsverfahren dargestellt (MA: Ergebnisse basieren auf Berechnung mit morphologisch bestimmten Taxalisten mit Abundanzwerten, M01: Ergebnisse basieren auf Berechnung mit morphologisch bestimmten Taxalisten mit An-/Abwesenheitsdaten, G: Ergebnisse basieren auf Berechnung mit genetisch erstellten Taxalisten mit An-/Abwesenheitsdaten). Statistisch hoch signifikante Korrelationen sind durch *** gekennzeichnet.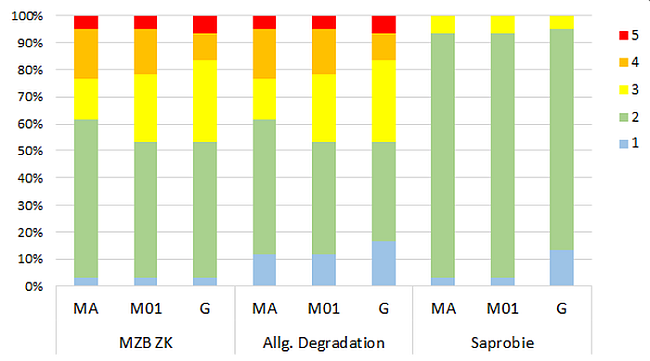 Abbildung 6: Verteilung der Bewertungsergebnisse (Zustandsklassen 1: sehr gut bis 5: schlecht) der unterschiedlichen Verfahren für die Gesamtbewertung der biologischen Qualitätskomponente Makrozoobenthos (MZB) und für die MZB Teilmodule "Allgemeine Degradation" und "Saprobie" (MA: Ergebnisse basieren auf Berechnung mit morphologisch bestimmten Taxalisten mit Abundanzwerten, M01: Ergebnisse basieren auf Berechnung mit morphologisch bestimmten Taxalisten mit An-/Abwesenheitsdaten, G: Ergebnisse basieren auf Berechnung mit genetisch erstellten Taxalisten mit An-/Abwesenheitsdaten).
Abbildung 6: Verteilung der Bewertungsergebnisse (Zustandsklassen 1: sehr gut bis 5: schlecht) der unterschiedlichen Verfahren für die Gesamtbewertung der biologischen Qualitätskomponente Makrozoobenthos (MZB) und für die MZB Teilmodule "Allgemeine Degradation" und "Saprobie" (MA: Ergebnisse basieren auf Berechnung mit morphologisch bestimmten Taxalisten mit Abundanzwerten, M01: Ergebnisse basieren auf Berechnung mit morphologisch bestimmten Taxalisten mit An-/Abwesenheitsdaten, G: Ergebnisse basieren auf Berechnung mit genetisch erstellten Taxalisten mit An-/Abwesenheitsdaten).Betrachtet man die Gesamtbewertung Makrozoobenthos (MZB ZK), erzielten die genetischen Verfahren mehrheitlich mit dem klassischen Verfahren vergleichbare Ergebnisse (63%). Bei 20% bewertete das genetischen Verfahren schlechter und bei 17% besser als das morphologische Verfahren (Abbildung 7). Diskrepanzen zwischen den Bewertungsergebnissen basieren unter anderem darauf, dass wichtige Indikatorarten der Plathelminthes, die Strudelwürmer (Turbellaria), nicht und einige wichtige Indikatorarten der Weichtiere (Mollusca) nur eingeschränkt genetisch detektiert werden konnten.
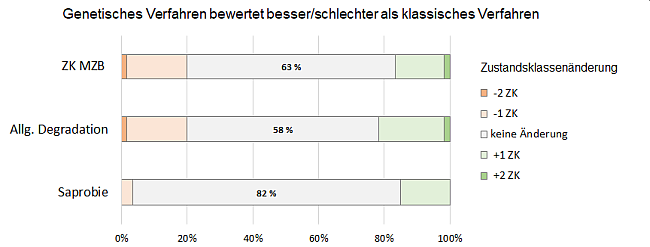 Abbildung 7: Darstellung der Zustandsklassenänderungen, die sich durch den Ersatz des klassischen Bewertungsverfahrens (Berechnungen mit morphologisch bestimmten Befunden inklusive Abundanzwerte) durch genetische Verfahren (Berechnungen mit genetisch ermittelten Befunden mit An-/Abwesenheitsdaten) für die biologische Qualitätskomponente Makrozoobenthos und die Teilmodule "Allgemeine Degradation" und "Saprobie" ergeben. In den Säulen sind die prozentualen Anteile der jeweiligen Zustandsklassenänderungen dargestellt.
Abbildung 7: Darstellung der Zustandsklassenänderungen, die sich durch den Ersatz des klassischen Bewertungsverfahrens (Berechnungen mit morphologisch bestimmten Befunden inklusive Abundanzwerte) durch genetische Verfahren (Berechnungen mit genetisch ermittelten Befunden mit An-/Abwesenheitsdaten) für die biologische Qualitätskomponente Makrozoobenthos und die Teilmodule "Allgemeine Degradation" und "Saprobie" ergeben. In den Säulen sind die prozentualen Anteile der jeweiligen Zustandsklassenänderungen dargestellt.Fazit – Was sind die Potentiale und Schwächen genetischer Verfahren?
Diese Studie hat gezeigt, dass genetische Verfahren das Potential haben, das klassische Verfahren basierend auf morphologischer Bestimmung für die biologische Qualitätskomponente Makrozoobenthos zu komplettieren bzw. zu ergänzen. Dies wird durch die wesentlich höher genetisch detektierte Artauflösung für einige morphologisch schwer bestimmbare Artgruppen, die hoch signifikanten Korrelationen der Bewertungsergebnisse und die mehrheitlich übereinstimmenden Zustandsklassen deutlich. Dennoch hat diese Studie auch gezeigt, dass genetische Verfahren das klassische Verfahren aufgrund deutlicher Abweichungen in den Taxalisten gegenwärtig nicht ersetzen können. Diese Abweichungen basieren auf einigen methodischen Schwächen der genetischen Verfahren, die zukünftig adressiert werden sollten, um die genetische Wiederfindung der Taxa in den Proben weiter zu verbessern. Ein für den Erfolg der Artwiederfindung essentieller und kritischer Arbeitsschritt ist die DNA Konservierung. Hier besteht die Notwendigkeit einer standardisierten und hinsichtlich DNA Konservierung optimierten Anleitung für die Probenahme und Handhabung. Um möglichst das gesamte Taxaspektrum der Probe genetisch abbilden zu können, muss die Wahl der entsprechenden Markergene und der Primer an die zu detektierenden Organismengruppen spezifisch angepasst werden. Die Integration von räumlichen Verbreitungsmustern der Taxa in die automatisierte taxonomische Zuordnung könnte die Genauigkeit der taxonomischen Zuordnung von genetisch sehr ähnlichen Taxa erhöhen. Eine hohe Qualität der genetischen Befunde sollte durch eine fortlaufende Optimierung und Aktualisierung der genetischen Referenzdatenbanken sichergestellt werden. Die durch genetische Verfahren erzielte höhere Artauflösung für spezifische Taxagruppen steigert zwar den Informationsgewinn, für die Gewässerbewertung ist diese höhere Artauflösung allerdings nur im Zusammenhang mit entsprechenden autökologischen Informationen gewinnbringend. Daher sind weitere Forschungsarbeiten hinsichtlich autökologischer Informationen und möglicher Indikatorpotentiale der genetisch detektierten Taxa sowie die entsprechende Anpassung der Taxaliste der Gewässerorganismen Deutschlands und der operationellen Taxaliste notwendig. Für die Integration genetischer Verfahren in das bestehende Bewertungsverfahren bedarf es einigen Anpassungen oder der Entwicklung neuer Bewertungsansätze (z.B. Ableitung neuer Metrics und Indizes, Strategie zum Umgang mit fehlenden Abundanzen).
Ausblick – was sind die nächsten Schritte?
Um weitere Erkenntnisse zu gewinnen und genetische Verfahren für die Anwendung in der behördlichen Praxis weiter zu optimieren, beteiligt sich das LfU neben dem Eco-Alps Water Projekt auch an dem UBA GeDNA-Projekt (Projektlaufzeit 2019 bis 2022) (gednaprojekt.wordpress.com). In diesem Projekt werden von der Universität Duisburg-Essen neben Makrozoobenthos- auch Phytobenthos Proben der Fließgewässertypen 5 und 9 (silikatische Mittelgebirgsbäche und –Flüsse) aus Sachsen, Nordrhein-Westfalen und Bayern genetisch analysiert. Zusätzlich wird auch die biologische Qualitätskomponente "Fischfauna" mittels Umwelt- oder environmental DNA (eDNA) untersucht. Neben dem Vergleich der Taxalisten und Bewertungsergebnisse sollen in dem GeDNA Projekt außerdem Korrekturfaktoren für die Gewässerbewertung aufgrund genetischer Erkenntnisse abgeleitet werden. Des Weiteren soll der genetisch basierte Erkenntnisgewinn interkalibriert und in die existierende Bewertungssoftware integriert werden.

